「超並列計算」可能なスーパーコンピュータでアミノ酸配列の相同性を高精度・高速検索 「PZLAST」公開

情報・システム研究機構 国立遺伝学研究所、理化学研究所、PEZY Computing(東京都)の共同研究グループは、公開中の膨大なゲノム断片から予測したアミノ酸配列データをもとに極めて高速かつ高精度にアミノ酸配列の相同性検索を可能とする Web サービス「PZLAST」を開発したと発表した。データを削減・圧縮することなく、超高速かつ高精度な配列相同性検索を可能とする世界で初めてのWebサービスとしている。
MIMD型プロセッサ活用で「超並列計算」、1回で最大10,000 個の配列を検索可能に

マイクロバイオーム研究の急速な進展に伴い、DDBJ※1などが運営する国際塩基配列データベースには、マイクロバイオームの主体となる微生物集団のゲノム断片配列(メタゲノム配列)データが加速度的に蓄積されつつある。なかでも「ショットガンメタゲノム配列データ」※2は、未同定、未培養な細菌がもつ未知の遺伝子情報を大量に含むことから「遺伝子の宝の山」とも言われているが、これらのデータはテラバイト(TB)を越える巨大なものであり、短時間で相同性を実施できる技術が存在していなかった。
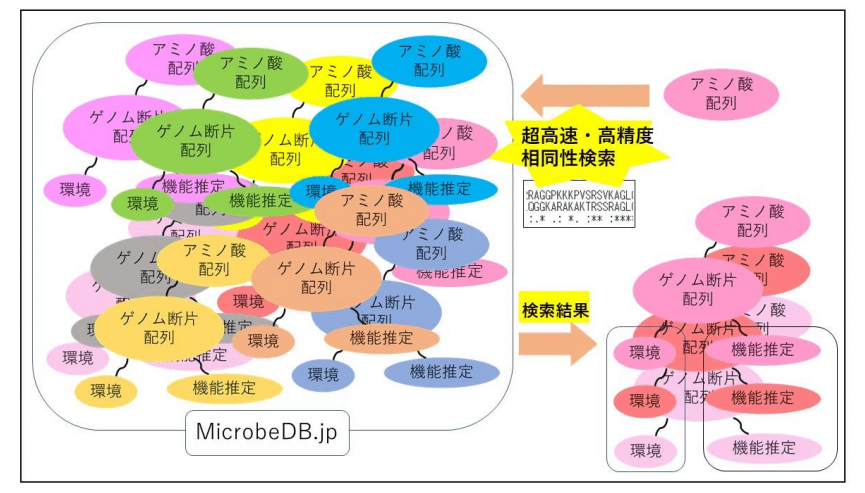
研究グループではこの課題を解決するため、PEZY Computing製のMIMD(Multiple Instruction Multiple Data)型メニーコアプロセッサー「PEZY-SC2」で構成される理化学研究所のスーパーコンピュータ「皐月」上で検索システムを構築した。MIMD 型プロセッサでは、各スレッドが異なるデータに対し異なる命令を実行可能となっており、これを活用すれば極めて多くの配列を同時に検索することが可能となる。今回開発したシステム「PZLAST」では、それぞれのPEZY-SC2において15,872 スレッドを利用する「超並列計算」が可能となり、極めて高速かつ高精度な配列相同性検索が実現したという。
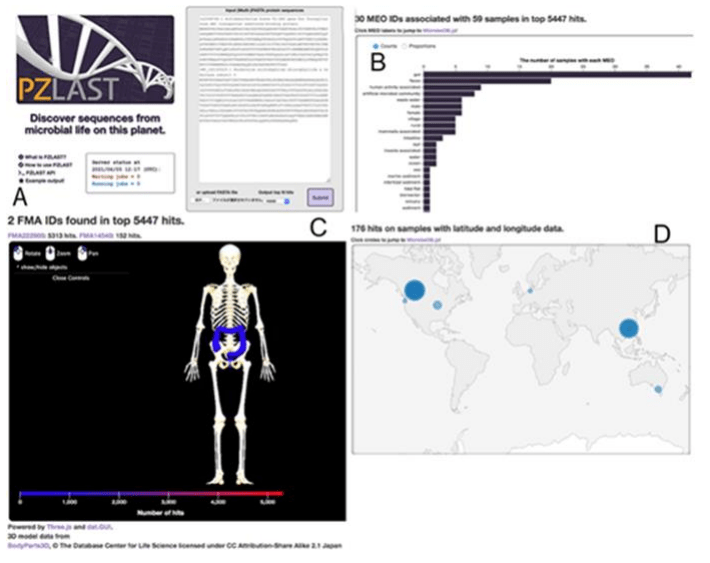
A. PZLAST ウェブサービスの検索クエリ入力画面。
B. 相同遺伝子の MEO による環境分布の例
C. 相同遺伝子の FMA によるヒトマイクロバイオームにおける分布の例
D. 相同遺伝子の地理的分布の例
PZLAST が相同性検索の際に参照配列として使用するアミノ酸配列データは、同じく国立遺伝学研究所で開発・運用している微生物統合データベース「MicrobeDB.jp」※3から取得する。MicrobeDB.jp では、DDBJから公開されているショットガンメタゲノム配列データから遺伝子を予測をするとともに、それら遺伝子の機能を推定した上で、サンプルが採取された「環境の情報」と統合してデータベースに格納している。すなわち、MicrobeDB.jp に格納されているすべての遺伝子データは、それら遺伝子がどのような環境中に存在したのかなど配列情報と環境情報が紐付けされているデータといえる。
PZLAST では、この MicrobeDB.jp から取得したアミノ酸配列を参照配列として相同性検索を行う。参照アミノ酸配列データは、4,339 サンプルのショットガンメタゲノムデータから予測された遺伝子群で、容量は約 2.5 テラバイト(423 億個の予測遺伝子数、アミノ酸約 1.7 兆残基)におよび「ヒト遺伝子 35 万人分」に相当する巨大なデータだという。ユーザーは 1 回の検索で最大 10,000 個の配列を入力することができ(図3A)、1回の検索に要する計算時間は約10分。検索結果は上位 1 万ヒットまでを、Metagenome and Microbes EnvironmentalOntology (MEO)クラス※4(図3B)、Foundational Model of Anatomy オントロジー(FMA)クラス※5(図 3C)、地理的分布(図 3D)に基づいてまとめ、Web ブラウザ上で可視化する。
研究グループでは、膨大なゲノム断片のデータが検索可能になったことで、薬剤耐性因子、病原因子やウイルスなど特定の遺伝子の環境中での動態や、新たな機能を持つ遺伝子の発見、遺伝子と環境の関係性の解析、創薬など、さまざまな研究の発展に寄与できるとしている。研究成果については国際計算生物学会誌「Bioinformatics」に 2021 年 7 月 8 日付で掲載された。
※1 DDBJ
DNA Data Bank of Japan。国立遺伝学研究所の生命情報・DDBJ センターが、欧州 EBI および米国 NCBI の 3局で国際塩基配列データベース連携 INSDC (International Nucleotide Sequence Database Collaboration) を組織し運用する塩基配列データベース。
※2 ショットガンメタゲノム解析
微生物群集のもつ遺伝情報を丸ごと解読する解析手法。
※3 MicrobeDB.jp
JST NBDC「統合化推進プログラム」において国立遺伝学研究所のゲノム進化研究室(黒川教授)が中心となり開発・運用を行なっている微生物の統合データベース (データベース URL: https://microbedb.jp )。
※4 MEOクラス
相同遺伝子の環境分布
※5 FMAクラス
相同遺伝子のヒトマイクロバイオームにおける分布
外部リンク:PZLAST
論文リンク:PZLAST: an ultra-fast amino acid sequence similarity search server against public metagenomes(Bioinformatics)





