がん病理組織画像の特徴をAIで数値化、ウェブサイトやアプリで公開 東京大ら
日本の研究グループが、がんの病理組織画像の特徴を数値化する人工知能(AI)を開発し、さらにその技術を実装したウェブシステムとスマートフォンアプリを公開した。アカウントを登録すれば世界中の誰もが使えるようになっており、発展途上国等の医療機関を含め、高価な機器を必要とせずに病理診断を行える環境を提供することで、病理診断の高度化・均てん化が期待できるとしている。
「バイリニア畳み込みニューラルネットワーク」技術投入で数値化に成功
東京大学 大学院医学系研究科 衛生学分野の河村大輔 助教、石川俊平 教授らの研究グループは、人体病理学・病理診断学分野の牛久哲男 教授、深山正久 教授(研究当時)、消化管外科学の瀬戸泰之 教授、東京大学医学部附属病院 免疫細胞治療学講座の垣見和宏 特任教授、日本大学医学部外科学系消化器外科学分野 山下裕玄 教授らのグループと共同で、深層ニューラルネットワーク※1を用いて、がん病理組織画像の組織学的特徴を数値化する技術を開発した。
通常、がんの診断は顕微鏡を用い病理組織像を観察する病理診断により行うが、基本的には個別の病理医の経験知に基づいている。これまで組織像の客観的な記載や数値化は難しいとされ、多くの症例情報の集積、他の臨床データとの定量的な比較、類似症例の検索、といったデータとしての有用性確立は道半ばの状況だ。そこで、研究チームはディープラーニング技術を用い、病理組織画像の特徴を捉える数値化技術の開発に取り組んだ。
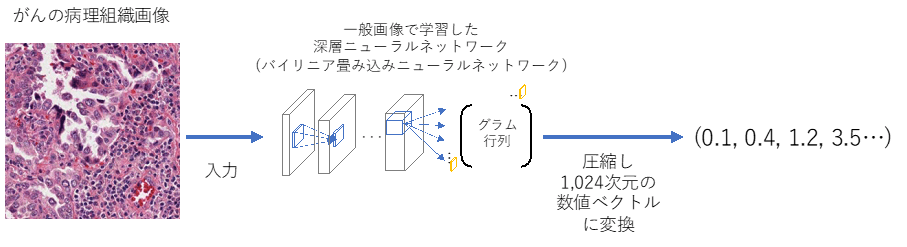
がんは単一の細胞を起源に持ち、同じ性質を持つ細胞が増殖する疾患であるため、がんの病理組織画像は明確な形状というよりはテクスチャのような特性を持つことが多い。そうした画像の解析に向いている「バイリニア畳み込みニューラルネットワーク」※4技術を採用し、病理組織画像をディープテクスチャ(図1)と呼ばれる1024次元の数値ベクトルに変換したところ、がんの組織学的特徴が極めてよく表現されることを発見したという。使用するニューラルネットワークの構造やパラメータによってその性能が異なるため、数値と病理医の評価を比較して検証を行った結果、病理組織像の評価に最適なネットワークと層の組み合わせを見出したとする。
新たな組織学的特徴も発見
研究グループでは、開発した技術の検証を以下3つのアプローチで検証、いずれも有意な結果を得ているとする。
①がん病理組織画像の組織学的特徴に基づく分類と可視化
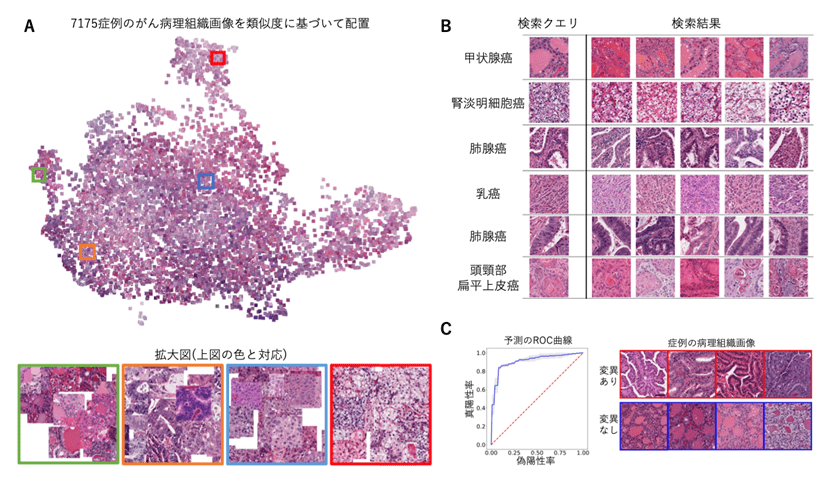
国際がんゲノムプロジェクト The Cancer Genome Atlas(TCGA)の32がん種7175症例の組織画像をその類似性に基づいて2次元に配置した結果、類似した形態を持つがん種同士が近くに配置されることを確認した(図2A)。サンプル数が少なく今後の検証は必要だが、胃がんにおいて免疫チェックポイント阻害薬の効果との関連を示唆する、これまで知られていなかった組織学的特徴を発見したという。
②類似症例検索
数値ベクトル同士の距離を計算することで、過去の症例と組織学的に類似した症例を高速かつ正確に検索することが可能となった(図2B)。病理医による評価との同等性も確認した。
③がん遺伝子変異の予測
今回の技術と一般的な教師あり機械学習技術を組み合わせることで、病理組織画像のみから309種類のがんと遺伝子変異の組み合わせが一定の精度で予測できることが示された(図2C)。
ウェブシステムとスマートフォンアプリを公開
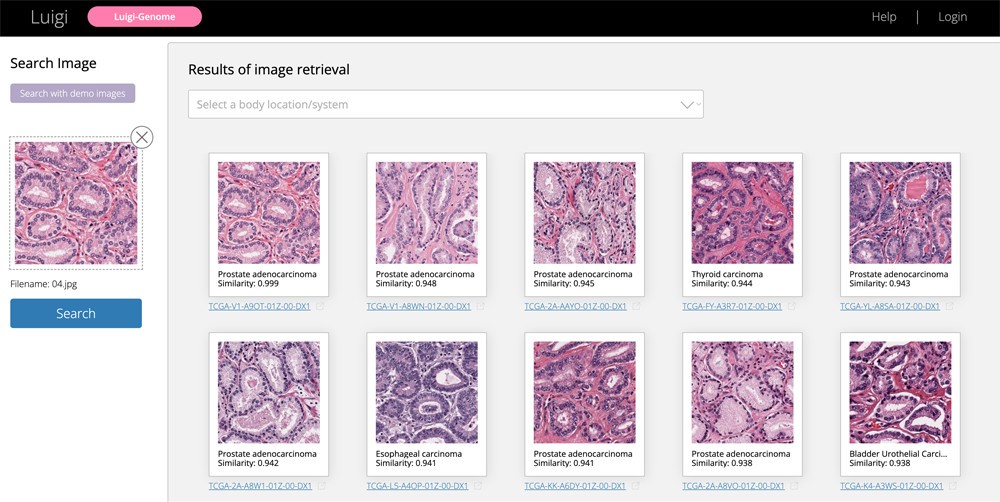
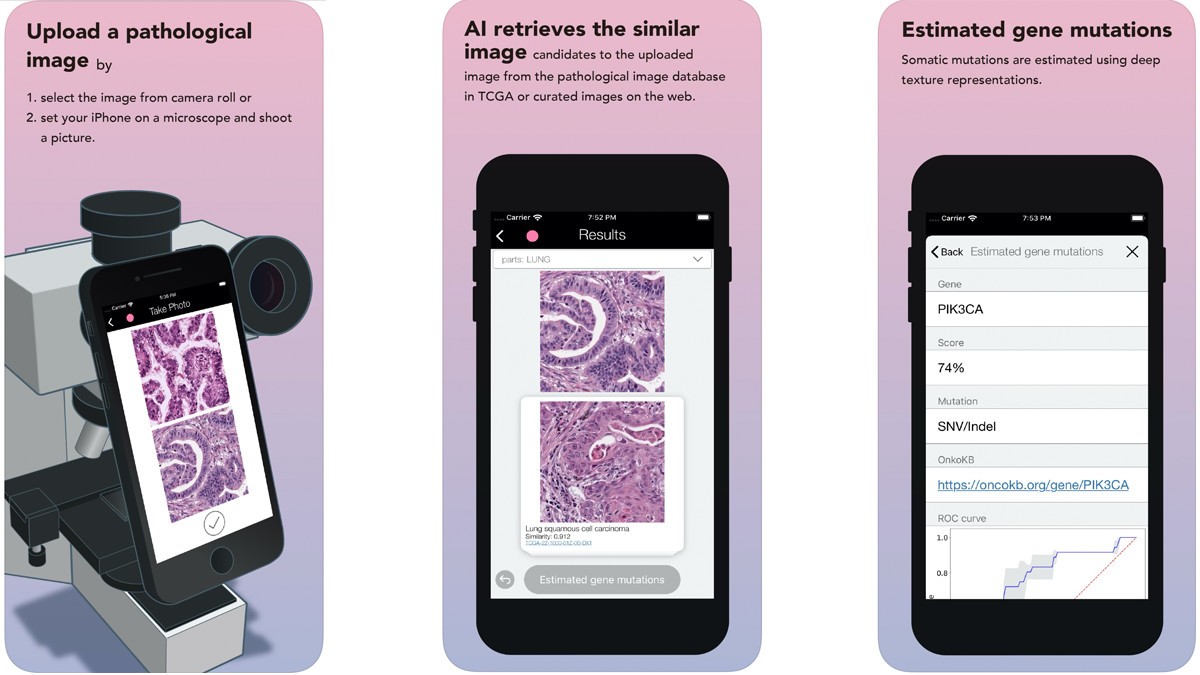
さらに、今回の技術のうち類似症例の検索機能および遺伝子変異の推定機能はウェブシステム(Luigi: Large Scale HistoPathological Image Retrieval System )やiPhone/iPad向けアプリケーション(App Store:検索ワード「luigi pathology」)として誰でも利用できるよう公開した。このツールは顕微鏡像をスマートフォン等で直接撮影もので動作するので、顕微鏡用デジカメシステム等の高価な機器を持たない中規模以下の医療機関や発展途上国の病院でも利用可能であり、病理診断の高度化・均てん化を促進できるとした。研究チームでは今後、さらなる精度の向上や類似症例検索のデータベースの拡充、医療機関での利用を想定した検証実験などを進めていく予定だ。なおこの成果についての論文は、2022年3月1日付で「Cell Reports」のオンライン版に掲載されている。
論文リンク:Universal Encoding of Pan-cancer Histology by Deep Texture Representations(Cell Reports)