日本の研究チーム、新しいゲノム編集ツール開発 筋ジストロフィー遺伝子修復に成功

京都大学iPS細胞研究所(CiRA)らの研究チームは、新たに大腸菌由来Type I-E CRISPRシステム(CRISPR-Cas3)を開発し、ヒト細胞でゲノム編集ツールとして利用できることを発見したと発表した。すでに研究の中で一部の筋ジストロフィー発現遺伝子の修復にも成功しており、さらなる研究進展、応用が期待される。
従来ツールより安全性向上
この新しい編集ツール「CRISPR-Cas3」を開発したのは、大阪大学医学部附属動物実験施設/東京大学医科学研究所の真下知士教授、大阪大学微生物病研究所の竹田潤二招へい教授、京都大学iPS細胞研究所(CiRA)の堀田秋津講師らの研究チーム。「CRISPR-Cas9」を代表とするゲノム編集ツールはさまざまなライフサイエンス分野に応用されていいる一方、既存技術は、狙っていない場所に変異が入るオフターゲット変異といった安全性への懸念や知的財産の問題がある。今回開発したCRISPR-Cas3は、狙ったゲノム配列の上流側を大きく削る性質を持ち、オフターゲットへの影響も極めて低いという。
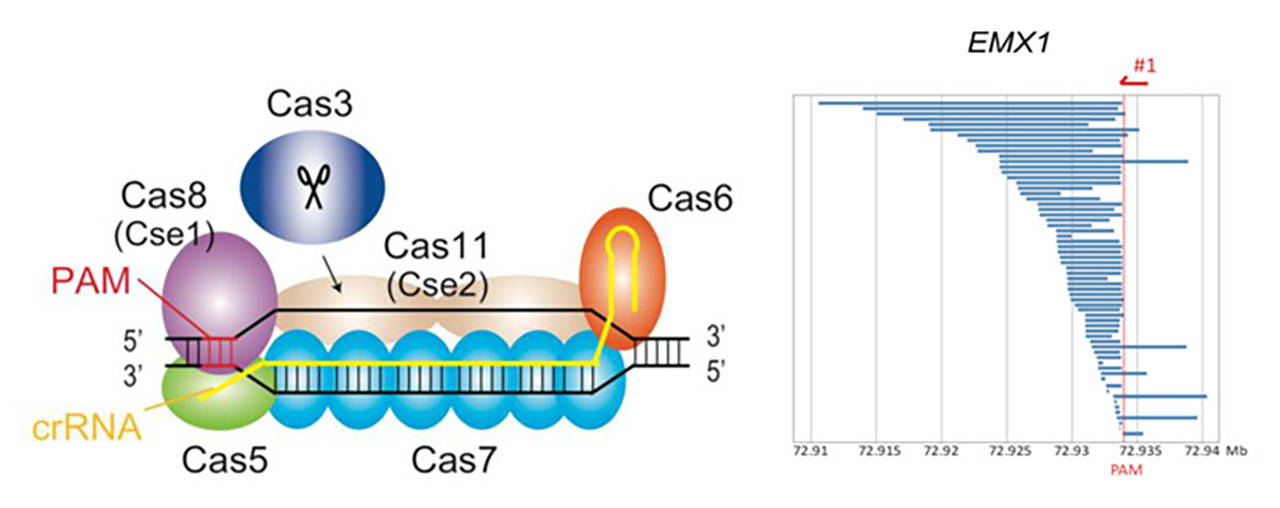
CRISPR-Cas3によって導入される切断パターンをキャプチャーシーケンスで解析したところ、標的配列の上流側に数百から数万塩基にわたって広範囲に配列が失われる欠失変異が導入されていることも判明した。この性質はCas9の標的部位に短い変異を導入する性質と大きく異なっており、Type I-E CRISPR-Cas3は、27塩基を標的として認識するcrRNAと5つの因子から成るCascade複合体がゲノム上の標的配列を認識して、その後Cas3がDNA切断を誘導する。懸念されるオフターゲットへの影響について、それぞれの技術で切断を誘導した全ゲノムシーケンス解析、また100カ所以上の類似領域のシーケンスを調べたところ、Cas9ではわずかにオフターゲット変異が確認された一方、Cas3ではオフターゲット変異は確認されなかった。すなわち、CRISPR-Cas3はオフターゲットへの影響がCas9と比較して少なく正確性が高いことが示唆された。
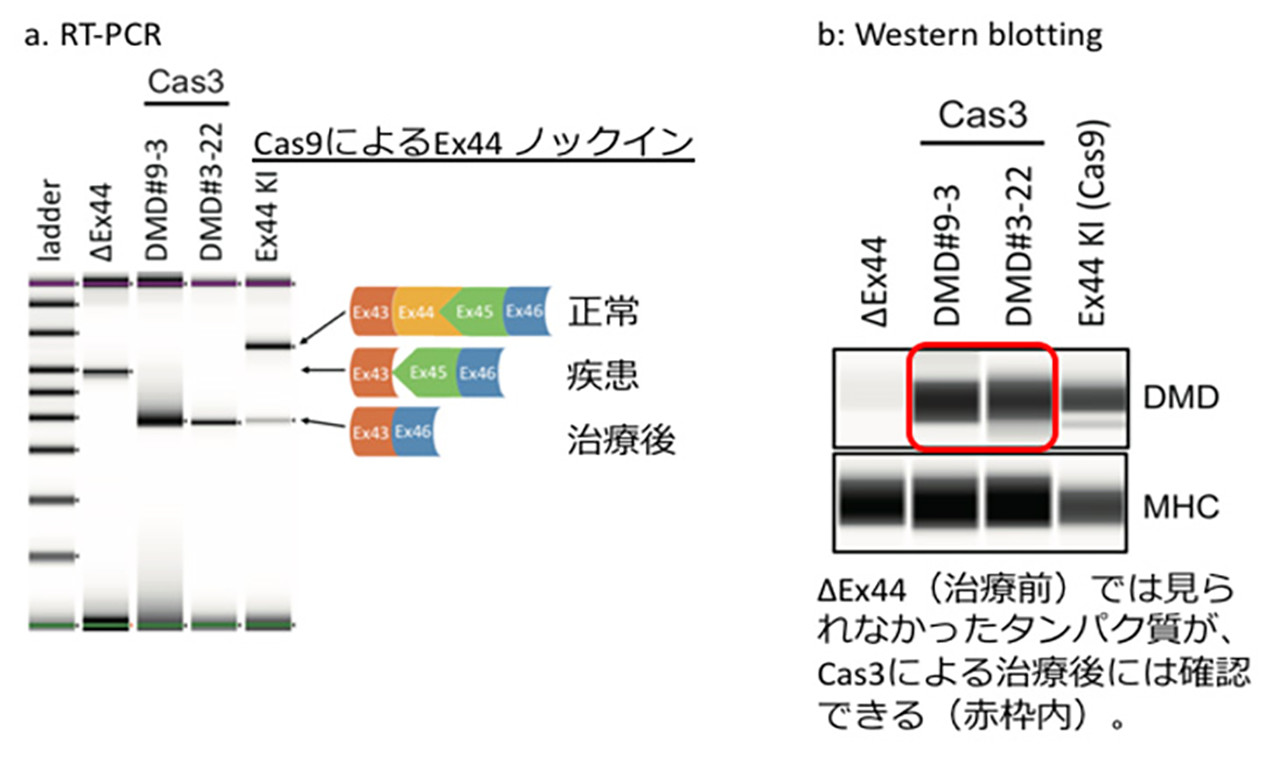
さらに、CRISPR-Cas3の遺伝子疾患に対する治療応用として、デュシェンヌ型筋ジストロフィー患者由来iPS細胞に利用した結果、エクソンスキッピング※1によりDMDタンパク質の発現が改善することを確認した。研究チームでは、CRISPR-Cas3はゲノムを大きく削ることから遺伝子や外来ウイルスなどを完全に破壊する、もしくは大規模領域をノックイン※2することに適していると考えられるという。また標的を認識する配列が、Cas9が20塩基に対してCas3は27塩基と多く、オフターゲットへの影響の少ない安全なゲノム編集基盤ツールと考えられるともしている。この本研究成果は、英国科学誌「Nature Communications」に12月6日付で掲載された。





